2023年3月18日,细胞生态海河实验室程涛教授牵头,联合基础、临床、生信平台等多个团队开展科研攻关,在《Science Immunology》(IF=30.63)期刊在线发表了题为“Single-cell dissection of human hematopoietic reconstitution after allogeneic hematopoietic stem cell transplantation”的研究论文(细胞生态海河实验室为第一完成单位)。首次刻画了患者经allo-HSCT后体内HSPCs和包括中性粒细胞各阶段在内所有成熟细胞的重建图谱,详细解析了HSPCs在allo-HSCT各阶段的精密调控,为应对allo-HSCT后可能出现的PGF、免疫重建差异和GVHD等提供了可靠的参考基准。
该研究利用了单细胞标记逆转录RNA测序法(STRT-seq)和10×Genomics平台的3’端单细胞转录组测序方法,分别对经流式分选后的Lin-CD34+细胞和总有核细胞(TNCs)进行建库及深度测序,并基于转录组特征对Lin-CD34+细胞分群:共分为HSC/多潜能祖细胞(MPPs),淋系启动的多能祖细胞(LMPPs),巨核红系祖细胞(MEPs),嗜酸-嗜碱-肥大细胞祖细胞(EBMs),B-NK祖细胞(BNKs),单能中性粒细胞祖细胞(NePs)和基质细胞(Stromal cells)等10群。首先分析了HSPCs在组分上的动力学变化,结果显示供者采集物(Donor)中HSC/MPPs和LMPPs比例显著增加,其占比在移植重建过程中逐渐下降;Donor和移植后第一天(d1)样本中HSPC组分更加相似,造血重建时(d14)和移植后30天(d30)骨髓(BM)中HSPCs构成相似;自移植后60天(d60)起,移植患者HSPCs组成开始出现差异,主要是BNKs所致。
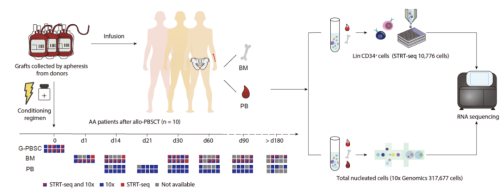
随后,该研究利用全外显子测序中的单核苷酸多样性信息,对6名移植患者移植后的BM/外周血(PB)细胞进行单细胞转录组嵌合分析。在随访中,患者遗留细胞集中于移植后第一天(d1),Lin-CD34+细胞中约有1%的细胞为受者遗留细胞,其中85.7%为基质细胞。在分析中发现了一例在移植后90天,遗留细胞骤增(PB中遗留T细胞占该时间点所有T细胞的36.2%)的特殊病例,该患者进行了全相合移植,且无急性移植物抗宿主病(aGVHD)发生,该病例可能代表了临床中一类极为特殊的患者群体,提示循环中的遗留T细胞可在移植患者体内复现并发挥潜在作用。
接下来,研究人员对HSC/MPPs、寡能祖细胞和中性粒细胞祖细胞进行详细阐释。研究发现,HSC/MPPs表现出巨核-红系偏倚和强烈的增殖倾向。与HSC/MPPs一致,d1捕获到的寡能祖细胞(MEPs、GMPs和EBMs)同样具有强烈的增殖倾向,并在转录组上表现为谱系分化受抑的模式。此外,该研究发现了具有独特免疫调控表达谱的粒系祖细胞——S100Ahigh Neus(包含S100Ahigh Neu1和S100Ahigh Neu2),其在粒细胞集落刺激因子(G-CSF)动员后富集,同时能够在移植患者的HSPCs中持续存在。前瞻性临床队列(n=32)提示,发生aGVHD的患者(n=17)的S100Ahigh Neu2输注量明显少于未发生aGVHD的患者(n=15,P<0.01)。
综上,该研究成果为移植后HSPCs的动力学重建提供了全面的见解和参考,为深入理解allo-HSCT的移植生态提供了宝贵的证据。同时,该研究提出了移植后免疫重建差异的时间窗口和识别aGVHD高危患者的早期预警指标,有助于临床诊疗中对移植患者发生重建不良及aGVHD进行早期诊断和干预,为基础研究向临床应用的转化提供了新角度。
细胞生态海河实验室程涛教授、郝莎副教授和姜尔烈教授为该文共同通讯作者。团队成员霍莹莹、武林杰、庞爱明、李清和洪芳为并列第一作者。
参考文献:
[1] Velardi, E., J.J. Tsai, and M.R.M. van den Brink, T cell regeneration after immunological injury. Nature Reviews Immunology, 2021. 21(5): p. 277-291.
[2] Waller, E.K., et al., Kinetics of immune cell reconstitution predict survival in allogeneic bone marrow and G-CSF–mobilized stem cell transplantation. Blood Advances, 2019. 3(15): p. 2250-2263.
[3] Biasco, L., et al., In Vivo Tracking of Human Hematopoiesis Reveals Patterns of Clonal Dynamics during Early and Steady-State Reconstitution Phases. Cell Stem Cell, 2016. 19(1): p. 107-119.
[4] Dong, F., et al., Differentiation of transplanted haematopoietic stem cells tracked by single-cell transcriptomic analysis. Nature Cell Biology, 2020. 22(6): p. 630-639.
[5] Xie, X., et al., Single-cell transcriptomic landscape of human blood cells. National Science Review, 2021. 8(3).
[6] Zheng, Z., et al., Uncovering the emergence of HSCs in the human fetal bone marrow by single-cell RNA-seq analysis. Cell Stem Cell, 2022. 29(11): p. 1562-1579.e7.
[6] Zhang, Y., et al., Temporal molecular program of human hematopoietic stem and progenitor cells after birth. Developmental Cell, 2022. 57(24): p. 2745-2760.e6.



